Plateforme de génomique et transcriptomique.
Séquençage et PCR digitale.
Conseil et prestation de service
Du contrôle qualité à l'analyse de données
Transfert de compétences
Ambassadeur Microbiologie au sein de la plateforme régionale GeT
Plus de 20 ans d'expérience
Veille technologique et scientifique
Collaboration et partenariat
Accord de confidentialité
Développement méthodologique selon vos besoins
Hébergement de projets
Pour le secteur public et privé
Local et international
Membre d'infrastructure de recherche IBISBA
Certifiée ISO-9001 et NFX50-900
Créée en 1999 et actuellement dirigée par Marie Ange Teste, la Plateforme technologique de recherche GeT-Biopuces est l'une des premières plateformes de la Génopôle Toulouse Midi Pyrénées. Elle est hébergée au sein du Toulouse Biotechnolgy Institute à l’INSA de Toulouse
Labellisée IBISA et certifiée ISO 9001 depuis 2010, la plateforme a une activité de prestation de services et/ou collaboration (R&D) dans le domaine de la transcriptomique et de la génomique. Elle met à disposition son expertise et possède les outils pour le séquençage, l’étude sur microarrays et l’analyse des données bioinformatiques et statistiques. Grâce au savoir-faire de son équipe pluridisciplinaire, vous êtes accompagnés tout au long de la réalisation de vos projets depuis leur conception jusqu'à la mise à disposition des données brutes ou traitées. De nombreux partenaires académiques (INRAE, CNRS, INSERM, IFREMER...) et privés nous ont fait confiance pour collaborer sur des projets de recherche fondamentale ou appliquée, dans des domaines variés tels que la microbiologie, les biotechnologies, l'agroalimentaire, la santé et l'environnement. Les questions de propriété intellectuelle et les accords de confidentialité peuvent être discutés avec le service juridique de l'INSA.
La plateforme GeT-Biopuces est membre des infrastructures nationale et européenne en biotechnologie, IBISBA-FR et IBISBA (Industrial Biotechnology Innovation and Synthetic Biology Accelerator). Elle est associée à trois autres sites toulousains pour former la plateforme GeT (Génome et Transcriptome de GenoToul). Ainsi, cette plateforme multisite vous apporte l’une des offres les plus complètes de France dans le domaine de la génomique et de la transcriptomique : les puces à ADN Agilent, le séquençage haut débit petits et longs fragments, le séquençage sur cellule unique, la transcriptomique spatiale, la PCR digitale, l’analyse de données bioinformatique et statistique.
Pourquoi travailler avec nous ?
La plateforme vous propose les prestations suivantes:
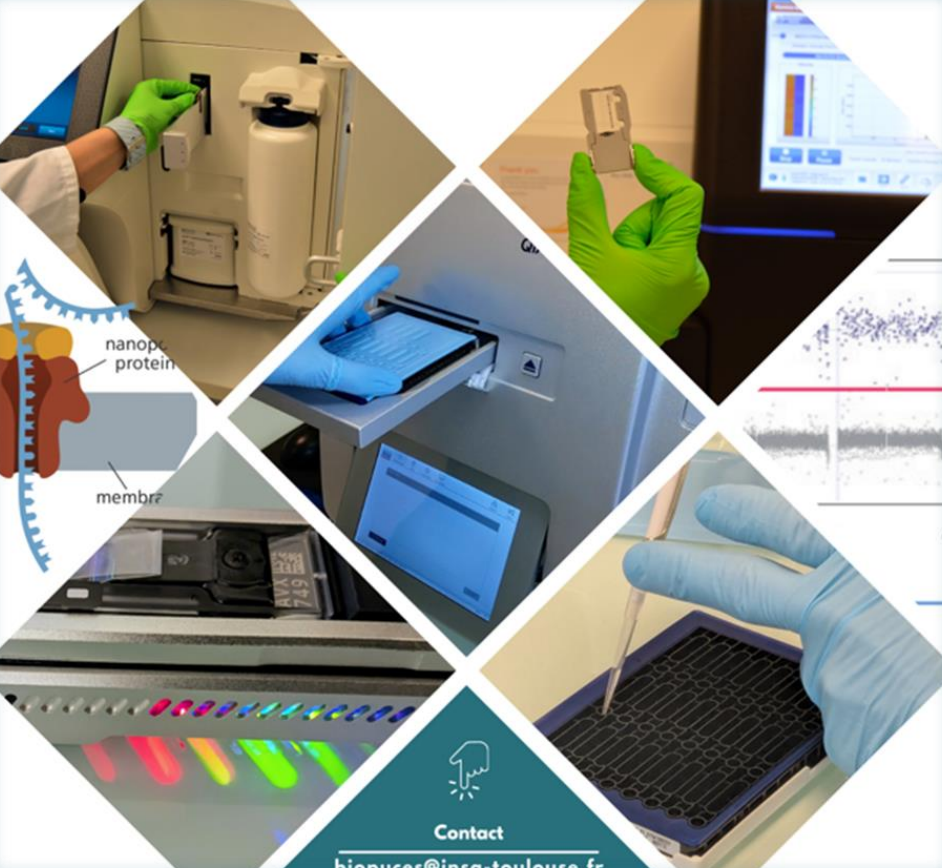
Bioanalyzer, Nanodrop, QuBit, SPECTROstar Nano
en savoir plus

Qiacuity Qiagen
en savoir plus

Ribodéplétion, Synthèse des librairies et séquençage
en savoir plus

Traitements bioinformatiques et analyses statistiques
en savoir plus
Pour tout renseignement sur l’une ou l’autre de nos activités, n’hésitez pas à nous contacter.
Pour en savoir plus sur notre façon de fonctionner, nous mettons à votre disposition nos recommandations (pdf).
 Télécharger (PDF)
Télécharger (PDF)
Ne sont ouvertes à la réservation directe que quelques machines.
Découvrez l'étendue des services offerts.
Toutes nécessitent une formation préalable.
Découvrez l'ensemble des équipements et offres de services de GeT.
Nous vous répondrons à get@genotoul.fr
Une équipe pluridisciplinaire répondra à vos demandes.







Plateforme GeT-Biopuces
TBI - INSA - Bât 39
135, avenue de Rangueil
31077 Toulouse Cedex 4 - France